Les bactéries peuvent être identifiées en utilisant la technique de biologie moléculaire PCR (Polymerase Chain Reaction). Une des stratégies possibles est d’utiliser la PCR pour amplifier un gène spécifique d’une bactérie recherchée, en utilisant des amorces s’hybridant sur ce gène et seulement ce gène. Le résultat sera alors de type « tout ou rien ». Une amplification signera la présence de la bactérie recherchée dans le produit analysé.
Les bactéries du genre Salmonella sont recherchées dans les produits biologiques en utilisant cette stratégie car elles sont responsables de gastro-entérites (salmonellose ; cf. scandale du lait Lactalis)
Ce type de détection est beaucoup plus rapide qu’une identification classique type galerie API 20 E et sérotypage (cf. TD « Identification et sérotypage »).
L’ARN 16S des bactéries du genre Salmonella semble leur être spécifique. Une paire d'amorces (« primers » en anglais) pour la détection PCR de Salmonella spp (espèces du genre Salmonella) a été conçu en utilisant comme cible le gène de l’ARN 16S.
Le produit attendu de la PCR, déterminé par bio-informatique, est un amplicon de 574 paires de bases..
L’objectif de l'activité est de vérifier que le gène de l’ARN 16S de Salmonella peut être exploité avec succès comme marqueur d’identification spécifiques des bactéries du genre Salmonella :
- Par la réalisation d’une technique de PCR dite « PCR sur colonie »
- Par la recherche des amplicons sur gel d’agarose après électrophorèse
Différentes souches bactériennes, dont une appartenant au genre Salmonella (bactérie de classe 2, maintenant plus utilisée par les élèves car tellement dangereux...) sont étaient disponibles dans le souchier du laboratoire.
Les colonies de ces souches sont mises en suspension et traitées avant PCR selon la procédure opératoire du document 1.
Le gène de l’ARN 16S est amplifié par PCR en utilisant deux amorces spécifiques (16S-sens et 16S-antisens) après plusieurs cycles de « dénaturation - hybridation des amorces- polymérisation » (Documents 2 et 3). Les produits d’amplifications obtenus sont analysés par électrophorèse en gel d’agarose (Documents 4 et 5).
- Emettre une hypothèse sur le rôle du traitement thermique des suspensions bactériennes.
- Compléter le tableau de manipulation proposé dans le document 3, pour cela :
- Repérer le volume final contenu dans le microtube.
- Calculer le volume de PCR Master Mix 2X à ajouter dans le microtube sachant qu’il doit être dans le volume final du microtube à une concentration 1X.
- Calculer le volume d’eau qualité BM (« Nucléase free ») à ajouter dans le microtube.
- Indiquer la composition qualitative et quantitative :
- d’un témoin négatif PCR
- d’un témoin de spécificité prouvant que les amplicons obtenus sont spécifiques de Salmonella.
- Schématiser un plan de dépôt en indiquant le sens de migration et le nom des électrodes.
- Prévoir les résultats à l’issue des manipulations en schématisant électrophorégramme attendu.
DOCUMENT 1 : traitement thermique des colonies bactériennes avant PCR (préparation de l’ADN matrice)
- Ajouter 200 µL d’eau qualité BM dans un microtube référencé au nom de la bactérie étudiée.
- Prélever un peu de la colonie correspondante à tester à l'aide d'un cône propre et stérile.
- La mettre en suspension dans le microtube contenant 200 µL d'eau qualité BM.
- Placer le tube contenant la suspension dans un bain thermostaté réglé à 95 °C durant 10 minutes.
- Conserver la solution dans la glace jusqu’au moment de la PCR.
DOCUMENT 2 : caractéristiques de la PCR sur colonie du gène sdiA
Séquence de l’amorce 16-Sens (forward) : 5' TGT TGT GGT TAA TAA CCG CA 3'
Séquence de l’amorce 16S antisens (reverse) : 5' CAC AAA TCC ATC TCT GGA 3'
L’amorce s’hybridant au brin matrice 3’ – 5’ s’appelle l’amorce sens (ou forward).
L’amorce s’hybridant au brin matrice 5’ – 3’ s’appelle l’amorce antisens (ou reverse).
La taille de l’amplicon est calculée avant la manipulation car la séquence à amplifier est connue. L’amplicon commence à l’extrémité 5’ de l’amorce sens et se termine à l’extrémité 5’ de l’amorce antisens.
Programmation du thermocycleur :
|
Cycles
|
Température
|
Durée
|
Nom de l’étape
|
|
Cycle 1
|
94 °C
|
5 min
|
Dénaturation initiale
|
|
Cycles 2 à 34
|
94 °C
|
30 secondes
|
Dénaturation
|
|
60°C
|
30 secondes
|
Hybridation des amorces
|
|
72 °C
|
4 min
|
Polymérisation
|
|
Cycle 35
|
72 °C
|
3 minutes
|
Polymérisation finale
|
|
Incubation
|
4°C
|
µ
|
Conservation avant dépôt
|
Taille du produit d’amplification : 574 paires de bases (pb)
DOCUMENT 3 : procédure opératoire de la réalisation des tubes PCR
|
Réactifs
|
Volumes à ajouter en µL
|
|
Volume PCR Master Mix 2X (Taq polymerase + dNTP)
|
12,5
|
|
Volume d’amorce 16S sens à 10 µmol · L-1
|
2,5
|
|
Volume d’amorce 16S antisens à 10 µmol · L-1
|
2,5
|
|
Volume de suspension bactérienne traitée pendant 10 minutes à 95 °C
|
2
|
|
Volume de MgCl2 à 25 mmol · L-1
|
2,5
|
|
Volume d’eau qualité BM qsp 25 µL
|
|
ATTENTION : préparer le mélange de PCR sur de la glace pilée dans un microtube. Lors du mélange et de l’homogénéisation, éviter de faire des bulles car celles-ci nuisent à la bonne réussite de la PCR
REALISATION PRATIQUE
T1. Préparation de l’ADN matrice
Réaliser l’extraction de l’ADN bactérien selon la procédure opératoire décrite dans le document 1.
T2 Amplification par polymérisation en chaine (PCR)
Réaliser l’amplification du gène de l’ARN 16S selon la procédure opératoire décrite dans le document 3.
T3. Electrophorèse sur gel d’agarose
Réaliser la recherche des amplicons par électrophorèse sur gel d’agarose.
RAPPORT D’ACTIVITE
- Annoter l’électrophorégramme en donnant : le titre, le sens de migration, la légende de la composition du contenu des puits et les conditions de migration.
- Analyser les témoins et conclure sur leur conformité respective.
Valider (ou non) la PCR réalisée.
- Comparer la taille des différents amplicons obtenus puis conclure.
- Valider que le gène de l’ARN 16S est bien caractéristique d’une part et spécifique d’autre part des bactéries du genre Salmonella.
Photo 1 : reconstitution des amorces designées pour amplifier le gène de l'ARN 16S

Photo 2 : fin de la migration (80 V, 1 h 30, gel à 1 % (m/V) d'agarose Topvision)

Photo 3 : avant révélation, on distingue les marqueurs colorés incorporées dans le Master Mix PCR 2X (rouge) et ceux incorporées dans le marqueur de taille "100 bp Plus DNA Ladder"

Photo 4 : résultats du scan avec le lecteur Geldoc EZ (Biorad) et révélation au FastBlast DNA Stain (marqueur de taille : "100 bp Plus DNA Ladder")
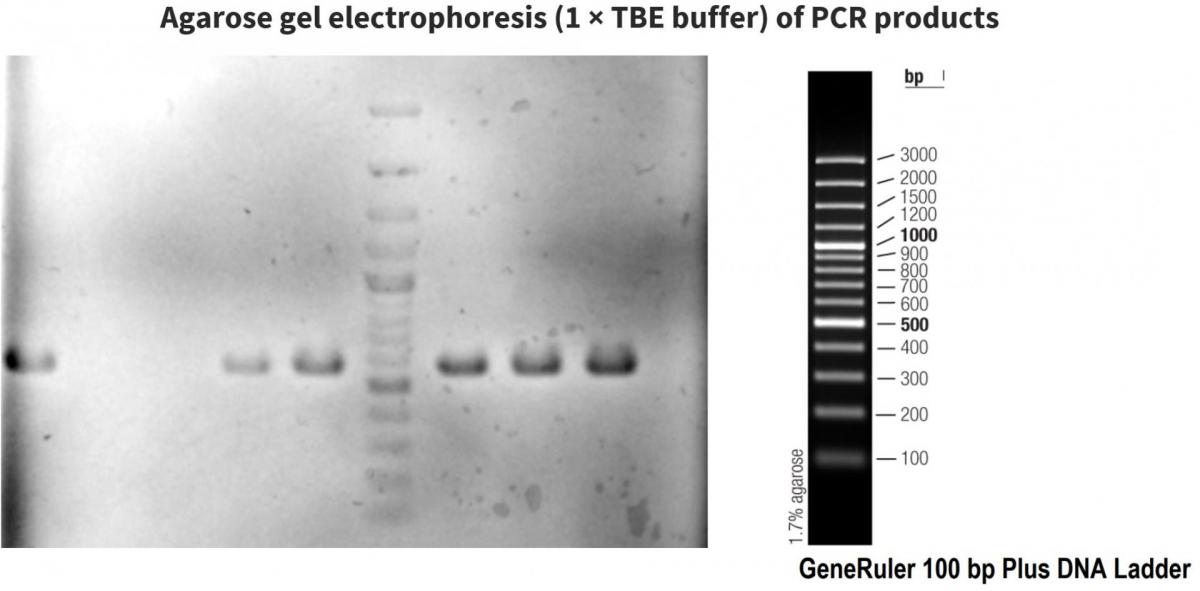
Puits 1 : Salmonella enterica spp enterica
Puits 2 : Escherichia coli
Puits 3 : Staphylococcus xylosus
Puits 4, 5, 7, 8, 9 : Salmonella enterica spp enterica
Puits 10 : Témoin négatif sans ADN
Puits 6 : marqueur de taille
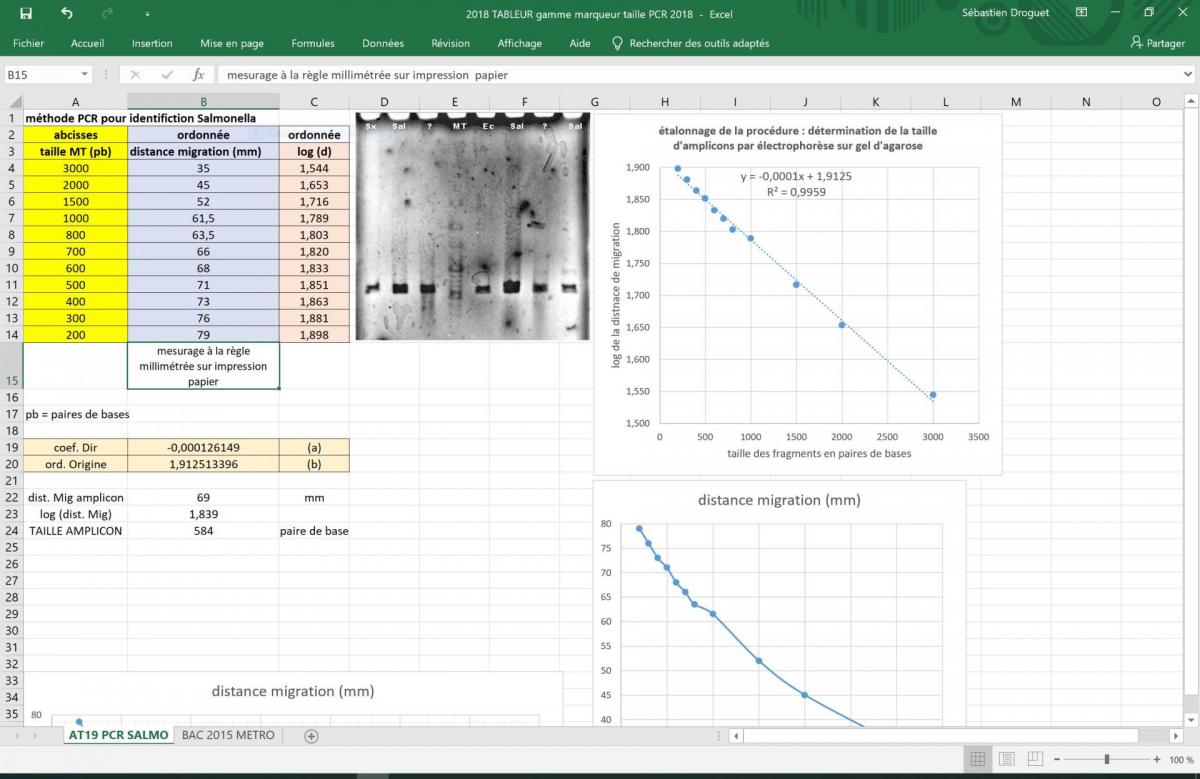
Exploitation de la gamme formée par le marqueur de taille "GeneRuler 100 bp DNA ladder"
CECI EST UNE DROITE REPRESENTANT L'ETALONNAGE D'UNE PROCEDURE ET NON UNE "COURBE DE CALIBRATION" COMME ON PEUT LE LIRE EN DIVERS ENDROITS, NOTAMMENT SUR UN SITE ACADEMIQUE
Article scientifique abordant ce thème : https://www.sciencedirect.com/science/article/pii/S1201971207000343
 Bonjour Sébastien
Bonjour Sébastien



