Objectifs :
- Etudier expérimentalement les propriétés spectrales particulières des acides nucléiques, liées à leur structure et composition.
- Quantifier avec exactitude et rapidité la concentration en masse de l'ADN en solution ρ(ADN ; solution ADN) .
La spectrophotométrie est le plus souvent utilisée car elle présente l’avantage de ne pas détruire l’échantillon.
Celui-ci peut alors être récupéré pour d’autres analyses et manipulations.
- Etudier les propriétés de solubilité de l'ADN
- réaliser une extraction d'ADN chromosomique bactérien et réinvestir les compétences précédemment acquises.
Utiliser Libmol pour visualiser la structure de la molécule d'ADN https://libmol.org
Libmol est une application en ligne, ergonomique et intuitive pour l'exploration des modèles de la librairie de molécules et d'ailleurs. Le site fonctionne avec tous les navigateurs modernes (Firefox, Chrome, Safari, Edge) et l’application existe en version téléchargeable
1) Onglet « fichier » è saisir « ADN » dans la case « rechercher dans la librairie de molécules »
2) Une portion de molécule d’ADN est disponible « ADN 14 paires de bases » è Cliquer dessus.
3) La molécule d’ADN modélisée en 3D peut subir rotations et agrandissement (roulette de la souris).
La signification des couleurs est indiquée en bas de la fenêtre.
4) Onglet « Commandes » è Plusieurs représentations possibles
Selon l’objectif voulu, certaines représentations seront plus adaptées que d’autres è à tester !
5) Onglet « Séquence » è se déplacer sur la séquence de la chaîne A ou B pour mettre en évidence les nucléotides concernés.
6) Partie supérieure droite : icones de capture d’image, plein écran, réglages divers à tester !
CHOIX DES CUVES DE MESURES ADAPTEES AU MESURAGE DE L'ABSORBANCE DANS LE BAS UV
Les cuves suivantes sont disponibles au laboratoire :

Réaliser, sur le même spectrophotomètre UV1600 PC et avec le logiciel MWave, les spectres d'absorption des cuves de mesures spectrophotométriques disponibles au laboratoire, dans le domaine de l'ultra-violet, en mode automatique (ajustage à zéro contre de l'air : porte-cuve vide).
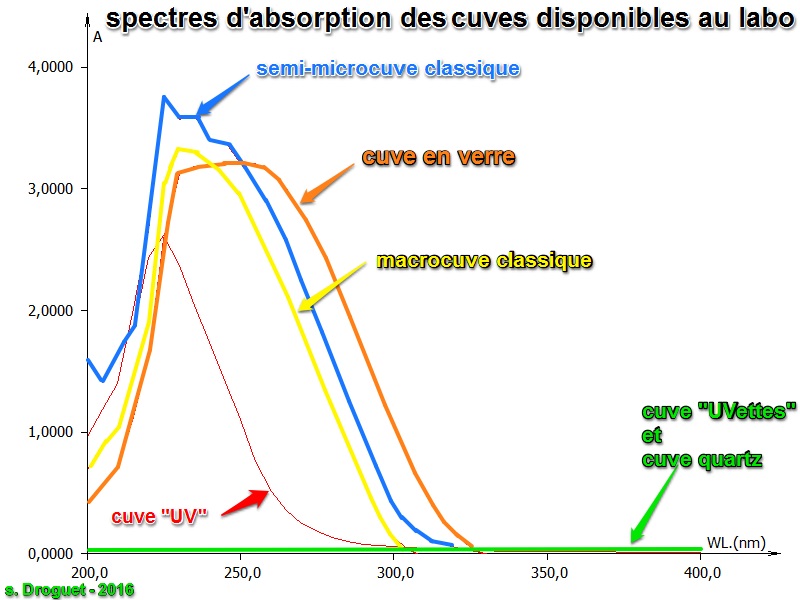
Présenter le document numérique regroupant l'ensemble des spectres réalisés.
Analyser et comparer les spectres d'absorption des cuves testées.
En déduire quelles sont la/les cuve(s) les plus adaptées à un dosage d'une molécule dans le domaine 200-300 nm.
Résumer trois critères de qualité d'une cuve dédiée à cet usage.
Remarque : deux instruments de mesure identiques (ici nos deux spectrophotomètres UV1600 PC) rendent toujours des indications différentes ; bien visible ici sur les spectres d'absorption de 4 cuves (chaque spectre ayant été réalisé sur les deux appareils avec exactement la même cuve) :


CHOIX DE LA LONGUEUR D'ONDE DE TRAVAIL POUR LE MESURAGE DE L'ABSORBANCE D'UNE SOLUTION D'ADN
Réaliser sur le même spectrophotomètre, dans le domaine [220 ; 300] nm, en utilisant la cuve de mesure adaptée, les spectres d'absorption des solutions suivantes :
- Solution d’ADN double brin de concentration inconnue (500 µL par cuve)
- Solution de protéine pure (Sérum-Albumine Bovine SAB) (500 µL par cuve)

Indiquer la composition du tube zéro permettant d’ajuster le spectrophotomètre avant la mesure de l’absorbance des solutions.
Présenter le document numérique regroupant les deux spectres réalisés.
Analyser et comparer les spectres d'absorption des solutions d'ADN et de protéine : déterminer, à l’aide de ces spectres, la longueur d’onde optimale d’absorption :
- d’une solution pure d’ADN ;
- d’une solution pure de protéine.
ANALYSE QUALITATIVE D'UNE SOLUTION D'ADN PAR SPECTROPHOTOMETRIE
Estimer, à l'aide des valeurs suivantes, la pureté d'une solution d'ADN fournie :
- vis-à-vis des protéines ;
- vis-à-vis des molécules organiques.
A260 nm = 0,805
A280 nm = 0,457
A230 nm = 0,379
Valider l’absence d’impuretés dans la solution.
|
Données :
Analyse du rapport R = A260 nm/A280 nm
Si 1,8 ≤ R ≤ 2 : purification satisfaisante
Si R ≤ 1,7 : les protéines n’ont pas été correctement éliminées
Si R ≥ 2 : les ARN sont en trop grande quantité
Analyse du rapport R = A260 nm/A230 nm
Si R < 2 : ADN contaminé par de l'EDTA, des glucides, du phénol..
Si 1,9 ≤ R ≤ 2,1 : ADN pur vis à vis d'autres molécules organiques
Si R ≥ 2 : les ARN sont en trop grande quantité
|
ANALYSE QUANTITATIVE D'UNE SOLUTION D'ADN PAR SPECTROPHOTOMETRIE
Ecrire, à l'aide des informations suivantes, la relation (basée sur la loi de Beer-Lambert, non exigée ici) entre l’absorbance d’une solution d’ADN bicaténaire et sa concentration.
|
Acide nucléique
|
Coefficient d'absorption linéique molaire
en mL ⋅ μg -1 ⋅ cm-1
|
Concentration en masse correspondant à une unité d’absorbance
|
|
ADN bicaténaire
|
0.020
|
rADNdb = 50 µg · mL-1
|
|
ARN monocaténaire
|
0.025
|
rARNsb = 40 µg · mL-1
|
En déduire la relation lorsqu’on travaille sur une solution diluée d’ADN.
Calculer ρ(ADN ; solution S) à partir de l'indication (absorbance) relevée sur le spectrophotomètre.
ETUDE DE L'EFFET DE LA CHALEUR SUR UNE SOLUTION D'ADN DOUBLE BRIN PAR SPECTROPHOTOMETRIE
Voici une série de mesurages d'absorbances :
| Sample Name |
Abs (260 nm) |
| 1 (25 °C) |
0,188 |
| 2 (25 °C) |
0,194 |
| 3 (25 °C) |
0,200 |
| 4 (25 °c) |
0,360 |
|
|
| 1 (80 °C) |
0,247 |
| 2 (80 °C) |
0,234 |
| 3 (80 °C) |
0,268 |
| 4 (80 °c) |
0,386 |
Analyser et comparer ces valeurs. Proposer une explication, en se basant sur les propriétés connues des nucléotides.
Mesurer l’évolution de l’absorbance à 260 nm d'une solution d'ADN double brin en fonction de la température notée θ en °C.
|
θ en °C
|
68
|
78
|
81
|
82
|
83
|
84
|
85
|
86
|
88
|
93
|
98
|
|
A260 nm
|
0,365
|
0,376
|
0,400
|
0,416
|
0,452
|
0,520
|
0,536
|
0,548
|
0,568
|
0,582
|
0,600
|
Une série de mesures d'absorbance à 260 nm (contreun blanc réactif adapté) d'une solution d'ADN chauffé à différente températures a permis de construire le graphique suivant :

Analyser ce graphe.
Utiliser la méthode des tangentes pour déterminer la température à laquelle la moitié des molécules d'ADN est dénaturée ( température de fusion ou Tm).
Etude de la solubilité de l'ADN (par Valérie Montalescot – Lycée de la vallée de Chevreuse – Gif sur Yvette) :
https://video.crdp.ac-versailles.fr/scolawebtv/3/1/2/54312.mp4
EXTRACTION D'ADN "CHROMOSOMIQUE" BACTERIEN
1. Lyse cellulaire et purification partielle de l’ADN
REACTIF : composition de la solution de lyse pour 600 µL
- 558 µL de tampon TN : Tris-EDTA 0,01 mol.L-1 NaCl 0,01 mol · L-1 pH 8
- 30 µL de SDS (= Sulfate de Dodécyl Sodium) à 10 % (m/V) - ATTENTION : pas de vortex, ça mousse avec le SDS
- 12 µL de solution de protéinase K à 20 mg.= · mL-1
- 5 mL de culture d’Escherichia coli (bouillon de 12 h) sont fournis : distribuer 1,5 à 2 mL dans un microtube (selon sa contenance) noté E et centrifuger à vitesse maximale.
- Eliminer délicatement le surnageant (pipetage). Répéter l’opération quatre fois de façon à culoter la totalité des 5 mL fournis..
- Ajouter au culot bactérien 600 µL de solution de lyse. Bien homogénéiser MAIS PAS AU VORTEX !
- Incuber 10 minutes à 50-60 °C.
- Centrifuger 5 min - 13 500 g (ou vitesse maximale) à 4 °C ATTENTION : solution TRES visqueuse !
- Prélever le surnageant avec une pipette à piston P1000 (couper la pointe du cône pour pouvoir pipeter) et le déposer dans un microtube annoté L+n° poste. Estimer le volume prélevé (graduations du microtube receveur ou utilisation de la pipette à piston).
2. Elimination de l’ARN par une ARNase
Le prélèvement est trop visqueux pour qu’une ARNase soit active.
Une utilisation ultérieure de cette enzyme sera envisageable (AT « extraction d’un plasmide »).
3. Précipitation de l’ADN chromosomique
- Ajouter 0,1 V d’acétate de sodium 3 mol.L-1 è ATTENTION : nécessite de bien avoir estimé Vsurnageant après le centrifugation précédente
- Ajouter 2 à 3 V d’éthanol 100 % glacé (conservé au congélateur) selon la contenance du microtube.
- Placer au moins 5 min à -20 °C (plus la durée est importante, meilleure sera la précipitation).
4. Elimination des sels
- Centrifuger 10 minutes à 4 °C à vitesse maximale.
- Eliminer le surnageant (voir paragraphe « sécurité ») dans le flacon "poubelle".
- Laver le culot sans le remettre en suspension en ajoutant 500 µL d’éthanol 70 % glacé (sorti du congélateur au dernier moment).
- Agiter le tube délicatement mais sans remettre l’ADN en suspension.
- Centrifuger 10 minutes à 4 °C à vitesse maximale.
- Eliminer le surnageant (voir paragraphe « sécurité ») dans le flacon "poubelle".
- Sécher le culot à l’étuve à 55 °C, microtube ouvert.
- Resolubiliser l’ADN obtenu dans 50 µL de TRIS-EDTA ou dans le tampon TN et le noter « A-N°poste ».
- Conserver dans la glace.
Sécurité :
- La croissance bactérienne mène à la formation de molécules acides qui ne sont pas compatibles avec l’eau de Javel (acides + Javel è dichlore néfaste pour les poumons) : il est donc nécessaire de jeter les surnageants de milieux de culture dans un tube qui sera autoclavé.
- Mettre le tube qui contient le culot de la première centrifugation (post lyse) dans un sac autoclave.
- Une fois les bactéries lysées, les déchets ne présentent plus de danger bio et peuvent être mis dans l’évier de par leur petit volume.
5. Analyse de l’ADN bactérien extrait par mesures spectrophotométriques
- Diluer au 1/10 le prélèvement « A-N°poste » dans un diluant adapté (tampon TN, TE ou eau qualité BM selon ce qui a été utilisé à la fin de l'étape 4) sous un volume final de 200 μL directement dans les microcuves adaptées.
- Mesurer l’absorbance de chacune des solutions diluées « A-N°poste - 1/10 » d’ADN à 260 nm et à 280 nm contre un blanc réactif adapté (tampon TN).
- Si l’absorbance est supérieure à 3,000, refaire une dilution plus importante sur le même volume final.
Définir le terme « ADNases ».
Relever les précautions mises en œuvre en biologie moléculaire pour empêcher la dégradation de l’ADN, molécule particulièrement fragile et sensible;
Rechercher le rôle des trois constituants de la solution de lyse utilisée pour l’extraction de l’ADN;
Expliquer la nécessité du chauffage à 60 °C.
Expliquer le rôle de l’acétate de sodium (CH3COO- Na+) vis-à-vis de la molécule d’ADN.
Expliquer le rôle de l’éthanol vis-à-vis de la molécule d’ADN
Calculer le rapport : A260 nm/A280 nm.
Conclure sur la pureté de l’ADN extrait.
Estimer la concentration de l’ADN extrait : établir l’équation aux grandeurs et l’équation aux unités puis faire l’application numérique.
Construire une synthèse de l'ensembles des conclusions obtenues