Objectif : réaliser un examen cytobactériologique (partiel) d'une urine incluant le dénombrement des germes urinaires, l'identification du micro-organisme responsable de l'infection et le choix de l'antibiothérapie à mettre en oeuvre.
Le diagnostic d’une infection urinaire est réalisé lors d’un ECBU (Examen Cytobactériologique des Urines) : examen de l’urine du patient réalisé après prélèvement d’urine vésicale dans des conditions optimales visant à minimiser les contaminations (désinfection soigneuse du méat urinaire, ensemencement immédiat des milieux de culture) et concentrer les éventuels micro-organismes présents (recueil de l’urine ayant séjourné au moins 3 heures dans la vessie) :
- examen macroscopique de l’urine : présence d’un trouble, de pus, de sang, couleur, odeur ;
- examen microscopique comprenant :
- un état frais : recherche/dénombrement d’hématies, de leucocytes (intacts, altérées ou en amas), de cellules épithéliales, de cylindres, de cristaux ;
- une coloration de Gram : pour la recherche de bactéries ;
- une coloration au bleu de méthylène : pour la recherche des éléments cellulaires ;
- une coloration de Ziehl éventuellement pour la recherche de mycobactéries.
- L’identification et le dénombrement des germes urinaires responsables de l’infection : sur milieu sélectif : gélose CLED, BCP pour des bactéries à Gram négatif, gélose au sang (milieu riche de par la présence de sang de mouton très riche en facteurs de croissance) si on observe des coques à Gram positif.
Dans la plupart des cas, une infection urinaire est monomicrobienne, mais des infections plurimicrobiennes sont possibles. À partir des isolements réalisés, on détecte le germe responsable de l’infection qui est celui présent en nombre significatif par rapport aux autres.
- Le profil de sensibilité d'une bactérie à différents antibiotiques par détermination de leurs concentrations minimales inhibitrices (CMI = plus petite concentration d'antibiotique suffisante pour inhiber de manière visible, au laboratoire, c'est-à-dire in vitro, la croissance d'une souche de bactéries après 24h à 37°C) :
- par dilution en milieu liquide ;
- par diffusion en milieu gélosé (antibiogramme standard).L'objectif est le suivant : un patient est atteint de fièvre, de douleurs lombaires aigues, accompagnées de douleur à la miction. Le laboratoire d’analyse doit effectuer le diagnostic de cette infection et choisir le traitement thérapeutique approprié.
1ère partie : dénombrement des germes urinaires (DGU) sur milieu CLED par la méthode à l'anse calibrée de 10 µL
Dénombrement réalisé directement à partir d'une "urine vésicale" sur milieu CLED.
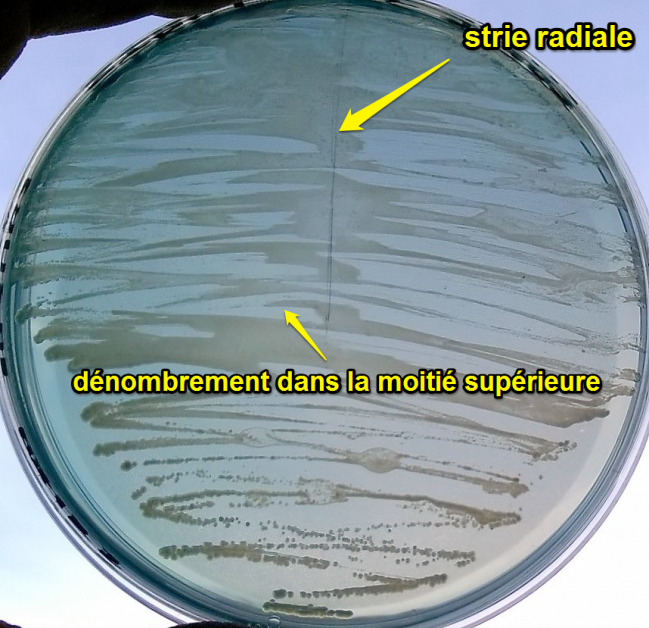
- Effectuer la lecture de la gélose CLED fournie. Interpréter la couleur des colonies (caractère lactose)
- Déterminer la bactériurie (nombre de bactéries·mL-1) de l’urine fournie en utilisant l’abaque suivant :
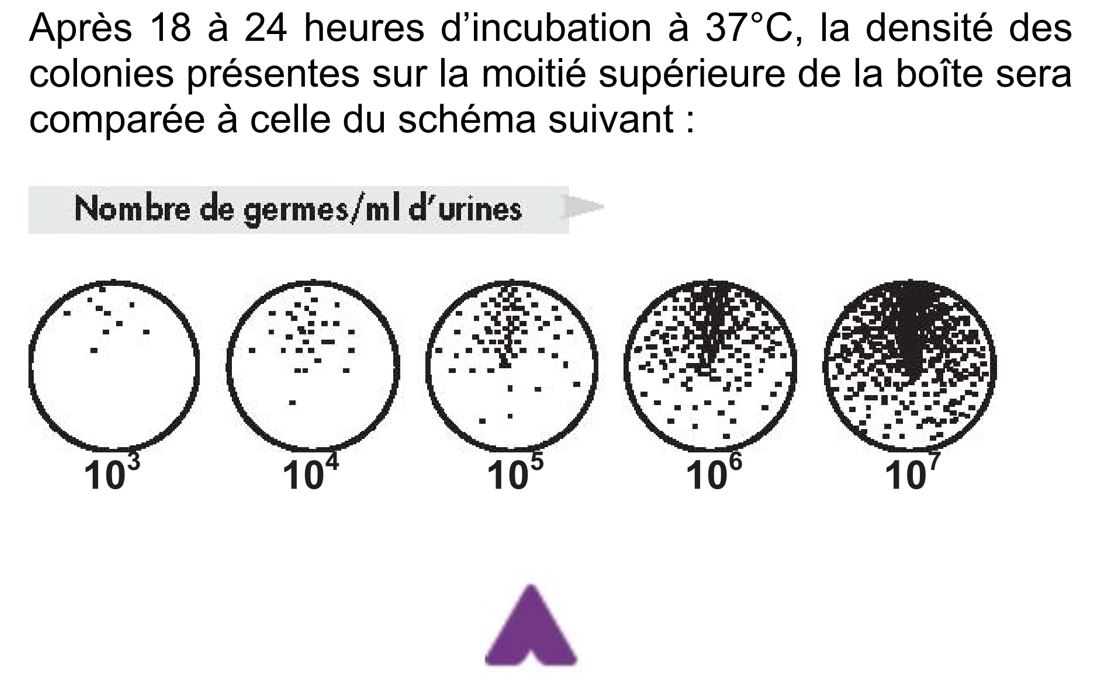

- Confirmer ou infirmer l’infection urinaire du patient. Justifier la réponse. Un examen complétentaire est-il nécessaire ?
2ème partie : orientation et identification du germe responsable de l'infection
Un isolement sur GNO a été réalisé à partir de l'urine précédente et fournie pour étude.
L'isolement montre un prélèvement monomicrobien, qui va servir à :
1) Préparer une suspension dense permettant de réaliser un frottis fixé coloré au Gram
2) Réaliser un test de discrimation rapide oxydase ou catalase en fonction du Gram
3) Ensemencer différents milieux :
- galerie de famille : Viande-Foie, Hugh & Leifson glucosé, Mannitol-Mobilité-Nitrates
- galerie "minimale" (réduite en raison du manque de certains milieux le jour de la manipulation) : Clark & Lubs, Citrate de Simmons, Hajna-Kliger

Voici les milieux obtenus après 24 h de culture à 37 °C
- Gram : bacilles roses, longueur 1,5 à 2 µm, isolés
- test oxydase : absence de coloration violette après dépôt d'une colonie sur le disque pré-imprégné de substrat NN diméthyl-paraphénylène-diamine (PDA
- Isolement sur GNO (fait en dernier, lu en premier) :

- Viande-Foie et Hugh & Leifson glucosé :

- Mannitol-Mobilité-Nitrates (ici après ajout des réactifs Nit1 et Nit2) :

- Hajna-Kligler :

Remarques :
- il faut ici vérifier la concordance du caractère lactose avec celui lu sur la gélose CLED
- un test enzymatique doit-être réalisé dans ce cas : le test oNPG
- Clark & Lubs :

Remarques :
- le test du VP apparait moins beau qu'au moment de sa lecture (photo tardive)
- le milieu citrate de Simmons n'est pas utilisable : en absence de milieu pour le préparer, une formule artisanale a été réalisée par la technicienne mais un composant de base faisait défaut.
Autre série de résultats, obtenus avec une souche bactérienne différente :
De gauche à droite : Viande-Foie, Hugh & Leifson, Citrate de Simmos, Kligler-Hajna, Mannitol-Mobilité-Nitrates
Gros plan sur une comparaison entre le citrate de Simmons inoculé et incubé (à gauche) et le même milieu non inoculé (à droite)
Gros plan sur Mannitol-Mobilité-Nitrates
Gros plan sur, de gauche à droite : test du VP, test du RM, test de l'oNPG, test de l'uréase
Gros plan sur la recherche de la production d'indole (à gauche) et de la recherche de l'activité TDA (tryptophane désaminase)
- Effectuer la lecture de la galerie de famille ; pour chaque milieu testé, présenter sous forme de tableau ; observation du résultat, interprétation et conclusion
- Confirmer la famille en argumentant.
- Effectuer la lecture de la galerie de genre/espèce : présenter, sous forme de tableau l’observation du résultat, l’interprétation et la conclusion.
- Indiquer, dans un premier temps, à quel groupe de genre appartient la bactérie à identifier.
- Identifier ensuite la souche à l’aide de tous les caractères obtenus par raisonnement dichotomique.
|
Famille ou
Genre
|
Famille Enterobacteriaceae
|
Genre Vibrio et
genres voisins
|
Genre Pseudomonas et
genres apparentés
|
|
Morphologie
|
Bacilles ou coccobacilles
|
Bacille fin, incurvés, 2-3 µm
|
Bacille fin droit
|
|
Groupement
|
Isolés
|
Isolés
|
Isolés / par 2
|
|
Gram
|
(-)
|
(-)
|
(+) ou (-)
|
|
Mobilité
ciliature
|
+ ou -
péritriche
|
++
Polaire
|
++
Polaire
|
|
Oxydase
|
(-)
|
(+)
|
(+) ou (-)
|
|
Voie d’attaque du glucose
|
fermentative
|
fermentative
|
Oxydative ou inerte
|
|
Exigeantes
|
non
|
non
|
non
|
|
Nitrate réductase
|
(+) stade nitrites
|
(+)
|
(+)
|
|
Type respiratoire
|
Aéro-anaérobie
|
Aéro-anaérobie
|
Aérobies stricts
|
|
Aspect en gélose
|
petites ou moyennes colonies (de 1 à 2 mm de diamètre pour 24 heures)
semi-bombées,
lisses,
arrondies
type smooth ; transparentes ou translucides
|
petites ou moyennes colonies (de 1 à 2 mm de diamètre pour 24 heures)
semi-bombées,
lisses,
arrondies
type smooth ;
transparentes ou translucides
|
pigmentées en bleu-vert, souvent plates, irrégulières,
s’étalant en 48h
|


3ème partie : détermination de la CMI de la ceftriaxone par détermination en milieu liquide (microméthode)
Ceftriaxone à 512 µg·mL-1 (CCI : < 4 µg·mL-1 et CCS > 32 µg/·mL-1)
|
Puits
|
1
|
2
|
3
|
4
|
5
|
6
|
7
|
8
|
9
|
10
|
11
|
12
|
|
VMueller
|
100 µL
|
100 µL
|
100 µL
|
100 µL
|
100 µL
|
100 µL
|
100 µL
|
100 µL
|
100 µL
|
100 µL
|
100 µL
|
100 µL
|
|
VAntibiotique
|
100 µL
|
|
|
|
|
|
|
|
|
|
|
/
|
|
Vredistribué
|
|
100 µL
|
100 µL
|
100 µL
|
100 µL
|
100 µL
|
100 µL
|
100 µL
|
100 µL
|
100 µL
|
100 µL
|
/
|
|
Vrejeté
|
/
|
/
|
/
|
/
|
/
|
/
|
/
|
/
|
/
|
/
|
100 µL
|
/
|
|
Vrestant
|
|
|
|
|
|
|
|
|
|
|
|
|
|
c(atb;tube)
µg·mL-1
|
|
|
|
|
|
|
|
|
|
|
|
|
|
Vinoculum
|
100 µL
|
100 µL
|
100 µL
|
100 µL
|
100 µL
|
100 µL
|
100 µL
|
100 µL
|
100 µL
|
100 µL
|
100 µL
|
100 µL
|
|
finale
µg·mL-1
|
|
|
|
|
|
|
|
|
|
|
|
|
|
Lecture
|
|
|
|
|
|
|
|
|
|
|
|
+
|

- Compléter le tableau de gamme
- Effectuer la lecture du témoin puis de la gamme.
- Déterminer la CMI de la ceftriaxone vis à vis de la souche testée (identification à faire en partie 2)
- Déterminer le profil de sensibilité de la bactérie vis à vis de la ceftriaxone
- Indiquer si cet antibiotique peut être utilisé pour traiter le patient.