L'AT précédente a permis de découvrir la notion d'orientation bactérienne, Dans le cadre d'un contrôle qualité au cours d'un processus impliquant une fermentation, deux contaminants bactériens ont été mis en évidence dans deux cuves distinces.
Dans chacun des cas, l'orientation a conduit à une bactérie de la famille des Enterobactericeae (ou Entérobactéries) : bacille, Gram négatif, oxydase (-), aéro-anaérobie, voie d'attaque fermentative du glucose, nitrate réductase (+) stade nitrites.
L’identification du genre et de l’espèce d’une souche bactérienne doit se poursuivre par la recherche de caractères biochimiques du métabolisme glucidique et protidique, en ensemençant une galerie d'identification : une "galerie" est un ensemble de milieux de culture, pouvant se présenter :
- soit en tubes (macro-galerie)
- soit en système miniaturisé (micro-galerie)

- L’objectif est de procéder à l'identification (genre, espèce) de chacun des deux contaminants, en comparant la méthode utilisant une macro-galerie classique (en tube) et une galerie miniaturisée.
1ère partie : réalisation d'un plan d'étude pour l'ensemcencement des milieux
Etape indispensable pour un ensemencement correct des milieux.
Le plan d'étude pour uen galerie API (ici API 10S) est fourni dans la notice du fabricant.
Le plan d'étude pour la macro-galerie est à réaliser avec la lecture des fiches techniques de chaque milieu (ici : Kligler-Hajna, Citrate de Simmons, Urée-tryptophane, Clark & Lubs, LDC, ODC)
2ème partie : ensemencement de la macro-galerie
Galerie complète après ensememencement, incubation et ajout des réactifs
Souche A : dans l'ordre :
1) Viande-Foie 2) Hugh & Leifson 3) Bouillon nitraté + nit1 + nit2 + poudre de zinc
4) Kligler-Hajna 5) Citrate de Simmons 6) Milieu de Falkow pour recherche ODC 7) Milieu de Falkow pour recherche LDC
8) Clark & Lubs 9) et 10) Urée-tryptophane

Souche B : mêmes milieux

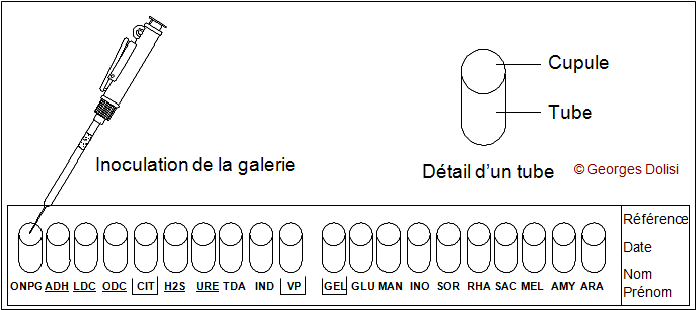
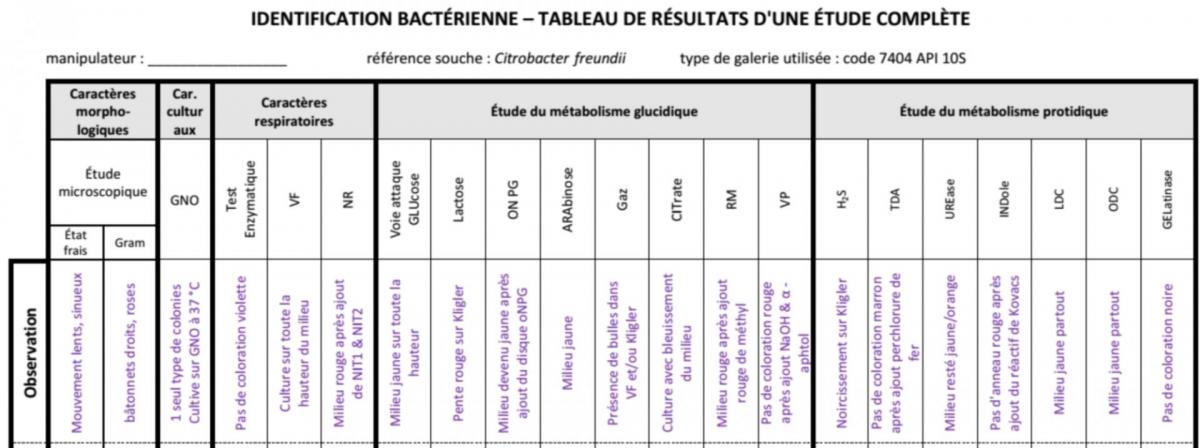
3ème partie : ensemencement de la macro-galerie : API 10S
La galerie API 10S (Biomérieux) est un galerie de 10 microtubes prêts à l’emploi contenant un susbtrat déshydraté, permettant de réaliser 10 tests biochimiques afin d’identifier des bacilles à Gram (–) appartenant à la famille des ENTEROBACTERIACEAE (c'est une version simplifiée de la galerie API 20 E : 10 tests au lieu de 20)
L'ensemencement se déroule de façon similiaire pour ces deux galeries :

Introduire la suspension bactérienne dans chaque tube à l’aide d’une pipette Pasteur stérile ouverte, pointe appuyée à l’intérieur et sur le côté pour éviter la formation de bulles (consigne importante à respecter).
La notice fait référence aux étalons de turbidité Mc Farland :

Etalons Mc Farland disponibles dans notre laboratoire (le plus couramment utilisé étant le "0,5"

Galerie après ensemencement avec une suspension d'opacité équivalente à un étalon de turbidité 0,5 Mc Farland, mais avant incubation : remplissage correct

Exemple de galerie API 10S après ensemencement , après incubation mais avant ajout des réactifs (dans puits TDA et IND) :
1) remplissage correct :
ATTENTION : ces résultats ne correspondent pas à ceux de la souche A de la souche B présentées ci-dessus

Remplissage incorrect (non respect des consignes)

Exemple de galeries après ensememencement, incubation et ajout des réactifs (dans chaque cas, l'orientation a conduit à une bactérie de la famille des Enterobacteriaceae)
Exemple 1 (souche A de l'orientation et de la macrogalerie)

Remarques : des gouttes sont sorties des tubes, traduisant un geste indélicat !
la galerie utilisée ici était périmée depuis quelques mois. Il a pu être constaté que certains résultats étaient faussement négatifs par comparaison avec la macro-galerie.
Exemple 2 (ne correspond ni à la souche A, ni à la souche B)

Exemple 3 (ne correspond ni à la souche A, ni à la souche B)

Tableau de lecture :

Explications concernant l'interpétation de la galerie API10S
oNPG : on recherche la présence de l'enzyme (ONPG hydrolase, équivalente en terme de réaction chimique à la bêta-galactosidase) par l'hydrolyse d'un substrat incolore (l'ortho nitrophényl galactoside) en produit coloré (chromophore) : le développement d'une coloration jaune traduit la présence de cette enzyme.
GLU : on recherche l'utilisation (par voie oxydative ou fermentative) du glucose (un aldohexose : ose à 6 carbones et fonction aldéhyde), se traduisant par une modification du pH du milieu faisant virer l'indicateur coloré de pH présent => métabolisme des glucides
ARA : même principe mais appliqué à l'arabinose (un aldopentose : ose à 5 carbones et fonction aldéhyde) => métabolisme des glucides
LDC: on recherche la présence d'une enzyme, la Lysine Décarboxylase, en mettant en évidence la décarboxylation (perte d'un carbone sous forme de CO2) de la Lysine (un acide aminé) en cadavérine ; l'alcalinisation du milieu est révélée par le virage d'un indicateur coloré de pH incorporé (pourpre de bromocrésol) => métabolisme des protides
ODC : on recherche la présence d'une enzyme, l'Ornithine Décarboxylase, en mettant en évidence la décarboxylation (perte d'un carbone) de l'Ornithine en Putréscine ; l'alcalinisation du milieu est révélée par le virage d'un indicateur coloré de pH incorporé (pourpre de bromocrésol) => métabolisme des protides
CIT : on recherche l'utilisation du citrate comme seule source de carbone (pour ce faire, il faut veiller à introduire aucune autre molécule organique pouvant servir de source de carbone). Une assimilation se traduira par une alcalinisation du lmlilieu, révélée par le virage de l'indicateur coloré de pH incorporé (bleu de bromothymol).
H2S : on recherche la production de sulfure d'hydrogène (à partir de thiosulfate présent dans le miieu), révélée par la combinaison du H2S formé avec des ions Fe3+ incorporés dans le milieu, sous forme d'un précipité noir.
URE : on recherche la présence d'une enzyme, l'uréase, en mettant en évidence l'hydrolyse de l'urée incoporée dans le milieu en produit alcalin (carbonate d'ammonium), se traduisant par une alcalinisation du milieu, révélée par le virage de l'indicateur coloré de pH incorporé (rouge de phénol).
TDA : on recherhe une enzyme, la Tryptophane Désaminase, en mettant en évidence la désamination du tryptophane en acide indole-pyruvique et NH3, révélé par un précipité brun caractéristique après ajout perchlorure de fer.
IND : on recherhe une enzyme, la tryptophanase, en mettant évidence le produit de l'hydrolyse du tryptophane, révélé par une coloration rouge caractéristique avec le réactif de Kovacs
OX : on recherche la présence d'une cytochrome oxydase (voir AT Orientation)
NO2 : on recherche la présence des nitrates réductase 1 et/ou 2 (voir voir AT Orientation)
Simulation de résultats tous négatifs avec l'identifieur en ligne de l'UPBM :

Simulation de résultats tous négatifs avec l'identifieur en ligne de l'UPBM :

4ème partie : identification du genre/espèc
1. Par approche dichotomique
les caractères sont hiérarchisés afin de construire un schéma d’identification que l’on suit pas à pas en fonction des résultats

Toutefois, le risque d'erreur est important par cette méthode ; la mauvaise lecture/interprétation d'un seul caractère peut mener à une mauvaise identification.
2. Par approche probabiliste utilisant un logiciel informatique
L'approche probabiliste est basée sur le principe que tous les caractères sont égaux (chaque caractère possède la même valeur discriminatrice, contrairement à l'approche dichotomique).
Une base de donnée (tableau, base de données en ligne ou incluse dans un tableur Excel) indique, pour chaque taxon et pour chaque caractère, sa probabilité d’être positif.
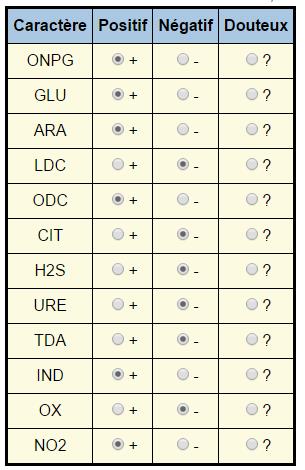
Voici un exemple de tableau (fourni avec les galeries API 10S) : chaque caractère testé la présence d'un chiffre qui indique le pourcentage de probabilité pour le caractère d'être positif

À l’aide de logiciels adaptés (exemple : le logiciel d'identification en ligne de l'UPBM., qui dispose d'une option "mode pédagogique" à cocher pour comprendre le fonctionnement de l'algorithme d'identification) on recherche le profil d’identification le plus proche de celui obtenu par la lecture de la galerie.
Le nom du micro-organisme est obtenu par un calcul de probabilité :
- à chaque caractère d’un micro-organisme donné fait référence une probabilité d’être (+) ou (-)
- la probabilité que ce soit un taxon donné correspond aux produits des probabilité attribué à chaque caractère
- le logiciel classe tous les résultats pour donner le ou les taxons les plus probables
En l'absence de logiciel, on peut aussi identifier manuellement, ligne par ligne, ce qui est fastidieux. Afin de gagner du temps, on peut appliquer la méthode suivante :
=> on utilise en premier les caractères dont on est certain et ceux qui sont positif
=> en l’absence d’autres caractères positifs, on utilisera les caractères négatifs
On procède ainsi :
- choisir un caractère (+) permettant de limiter la liste de suspects (10 à 12 si possible ; sinon, grouper 2 caractères positifs)
- relever le nom de chaque bactérie suspecte, et lui apposer un numéro (pour éviter de ré-écrire leurs nomtrs à chaque étape)
- prendre un autre caractère positif, et
> rechercher puis éliminer dans la liste les suspects (numéros) négatifs pour le caractère considéré
> rechercher puis retenir dans la liste les suspects (numéros) positifs pour le caractère considéré
- procéder ainsi pour tous les caractère positifs ;
- si un seul nom de genre/espèce apparait, vérifier que tous les autres caractères (négatifs) sont concordants ;
- si plusieurs noms de genre/espèce apparaissent, utliser alors les caractères négatifs.
Exemple pour la galerie 2 :
On prend les deux caractères CIT (+) et IND (+) pour limiter le nombre de suspects :
(1) Citrobacter koseri/amalonaticus
(2) Klebsiella oxytoca
(3) Pantoea spp2
(4) Proteus vulgaris
(5) Providencia rettgeri
(6) Providencia stuartii
(7) Serratia odorifera
(8) Aeromonas hydrophila
(9) Vibrio alginolyticus/parahaemolyticus
(10) Vibrio vulnificus/cholerae
Remarque : on peut directement éliminer (9) et (10) sur la base de la morphologie observée au Gram (bacilles droits pour la bactérie de cet exemple et non vibrion)
On prend le caractère LDC (+) :
(1), (3), (4), (5) et (6) sont éliminés car LDC (-)
Restent (2), (7) et (8)
On constate que ONPG (+), GLU (+) et ARA(+) ne sont pas des caractères dsiscriminants.
On prend le caractère ODC (+) :
(2) et (8) sont éliminés car ODC (-)
Reste (7) : Serratia odorifera
On vérifie la concordance de tous les autes résultats qui sont (-) : H2S (-), URE (-), TDA (-) : ils sont bien négatifs chez Serratia odorifera, qui serait donc la bactérie impliquée comme contaminant.
3. Par utilisation du codage API (profil numérique)
Sur la fiche de résultats, les tests sont séparés par groupes de trois et une valeur 1, 2 ou 4 est indiquée pour chacun.
- si le test est positif, lui attribuer la valeur indiquée dans la case (1, 2 ou 4)
- si le test est négatif, lui attribuer la valeur "0"
La galerie API 10 S comportant 10 tests, en additionnant à l'intérieur de chaque groupe les valeurs correspondant à des réactions positives, un profil numérique à 4 chiffres est obtenu.µ
La réaction de l'oxydase constitue le 11ème test et la réduction des nitrates en nitrites (NO2-) le 12ème test.
Exemple (ne correspond ni à la souche A, ni à la souche B de l'orientation et de la macrogalerie)

On peut soit utiliser le catalogue de profils fourni dans la notice :


soit utiliser ce site : http://210.242.211.31/servlet/Identify?action=prepareNew&stripId=23
soit utiliser ce site : http://lab.upbm.org/identifieur/
Exemple avec la bactérie 2 (identifiée ci-dessus) :

Avec la souche A (attention : certains caractères faussement négatifs sur API et le milieu Urée-Tryptophane n'avait pas été suffisamment ensemencé)

Avec la souche B (mêmes remarques que ci-dessus)

Il faut donc bien comprendre qu'il est indispensable de passer à une approche statistique globale (au lieu du dichotomique) ; le calcul ne donne pas une identité certaine mais seulement une probabilité d'identité, idéalement en proposant 2-3 taxons assortis de leur % de probabilité et en montrant que lorsqu'un taxon est très majoritairement probable c'est lui qu'on retient.
Identification obtenue pour la souche A
Raisonnement dichitomique fait par les élèves ==> Citrobacter farmeri, E. coli 2
Code numérique (avec le catalogue ou ici avec le site http://210.242.211.31/servlet/Identify?action=prepareNew&stripId=23)


Approche probabiliste (avec le site http://bioeluard.free.fr/api/)

Identification obtenue pour la souche B
A faire d'après les observations suivantes :
En conclusion (propos d'Antoine Gaudin)
On constate donc que, pour une espèce donnée, certains caractères ne sont pas soit totalement positif soit totalement négatif ; cette approche permet de comprendre que la diversité phénotypique (ce que l'on observe) est le support de l'identification, et que la démarche d'identification repose sur un départ dichotomique (Gram+/-, coque/bacille, prélèvement et symptômes...) mais montre rapidement ses limites (hiérarchisation des informations, erreur rédhibitoire, impasses) : on suit un chemin (pour reprendre les propos d'un collègue) mais il faut réfléchir à ce que l'on fait.
Sur le plan scientifique, ne faire que du dichotomique sur une galerie c'est n'avoir rien compris à l'identification et encore moins aux bactéries : les bactéries ne sont ni (+) ni (-) et ce parfois même sur des caractéristiques "fondamentales". Les tableaux api le montrent bien, avec leur % de positivité : les bactéries ont une probabilité d'être (+) ou (-).
L'objectif de la démarche globale est donc :
- pré orientation rapide dichotomique pour : sélectionner les milieux / la galerie idoine(s)
- galerie pour une collecte large d'infos phénotypiques ET traitement statistique de l'ensemble des infos collectées pour arriver à une identité "probable" (mais pas certaine : tenir compte des autres infos - par exemple une espèce voisine mais non pathogène ne peut pas être responsable des symptômes typiques)
S'entrainer à l'identification par les trois méthodes présentées à l'aide des galeries Exempes 1, 2 et 3 :


